Évaluation des modèles et d’ajustement des hyperparamètres
CSI 4506 - Automne 2024
Version: oct. 5, 2024 13h02
Préambule
Citation du jour
Objectifs d’apprentissage
- Comprendre l’objectif de la division des données :
- Décrire les rôles des ensembles d’entraînement, de validation et de test dans l’évaluation des modèles.
- Expliquer pourquoi et comment les ensembles de données sont divisés pour un apprentissage et une évaluation efficaces des modèles.
- Expliquer les techniques de validation croisée :
- Définir la validation croisée et son importance dans l’évaluation des modèles.
- Illustrer le processus de validation croisée \(k\)-fold et ses avantages par rapport à une seule division entraînement-test.
- Discuter des concepts de sur-apprentissage et de sous-apprentissage dans le contexte de la validation croisée.
- Ajustement des hyperparamètres :
- Expliquer la différence entre les paramètres de modèle et les hyperparamètres.
- Décrire les méthodes de réglage des hyperparamètres, y compris la recherche en grille et la recherche aléatoire.
- Implémenter l’ajustement des hyperparamètres à l’aide de
GridSearchCVdans scikit-learn.
- Évaluer la performance du modèle :
- Interpréter les résultats de la validation croisée et comprendre des métriques telles que la moyenne et l’écart-type des scores.
- Discuter de la manière dont la validation croisée aide à évaluer la généralisation du modèle et à réduire la variabilité.
- Flux de travail de l’ingénierie en apprentissage automatique :
- Décrire les étapes impliquées dans la préparation des données pour les modèles d’apprentissage automatique.
- Utiliser les pipelines scikit-learn pour un prétraitement efficace des données et un entraînement des modèles.
- Insister sur l’importance des transformations de données cohérentes entre les environnements d’entraînement et de production.
- Évaluation critique des modèles d’apprentissage automatique :
- Évaluer les limites et les défis liés à l’ajustement des hyperparamètres et à la sélection de modèles.
- Reconnaître les pièges potentiels dans le prétraitement des données, tels qu’une gestion incorrecte des valeurs manquantes ou un encodage incohérent.
- Plaider en faveur de tests et validations approfondis pour garantir la fiabilité et la généralisabilité du modèle.
- Intégration des connaissances dans des applications pratiques :
- Appliquer les concepts appris à des ensembles de données réels (par exemple, ensembles de données OpenML tels que ‘diabetes’ et ‘adult’).
- Interpréter et analyser les résultats des évaluations de modèles et des expériences.
- Développer une compréhension complète du pipeline d’apprentissage automatique de bout en bout.
Introduction
Ensemble de données - openml
OpenML est une plateforme ouverte pour partager des ensembles de données, des algorithmes et des expériences - pour apprendre à mieux apprendre, ensemble.
Ensemble de données - openml
Author: Vincent Sigillito
Source: Obtained from UCI
Please cite: UCI citation policy
Title: Pima Indians Diabetes Database
Sources:
- Original owners: National Institute of Diabetes and Digestive and Kidney Diseases
- Donor of database: Vincent Sigillito ([email protected]) Research Center, RMI Group Leader Applied Physics Laboratory The Johns Hopkins University Johns Hopkins Road Laurel, MD 20707 (301) 953-6231
- Date received: 9 May 1990
Past Usage:
Smith,J.W., Everhart,J.E., Dickson,W.C., Knowler,W.C., & Johannes,R.S. (1988). Using the ADAP learning algorithm to forecast the onset of diabetes mellitus. In {it Proceedings of the Symposium on Computer Applications and Medical Care} (pp. 261–265). IEEE Computer Society Press.
The diagnostic, binary-valued variable investigated is whether the patient shows signs of diabetes according to World Health Organization criteria (i.e., if the 2 hour post-load plasma glucose was at least 200 mg/dl at any survey examination or if found during routine medical care). The population lives near Phoenix, Arizona, USA.
Results: Their ADAP algorithm makes a real-valued prediction between 0 and 1. This was transformed into a binary decision using a cutoff of 0.448. Using 576 training instances, the sensitivity and specificity of their algorithm was 76% on the remaining 192 instances.
Relevant Information: Several constraints were placed on the selection of these instances from a larger database. In particular, all patients here are females at least 21 years old of Pima Indian heritage. ADAP is an adaptive learning routine that generates and executes digital analogs of perceptron-like devices. It is a unique algorithm; see the paper for details.
Number of Instances: 768
Number of Attributes: 8 plus class
For Each Attribute: (all numeric-valued)
- Number of times pregnant
- Plasma glucose concentration a 2 hours in an oral glucose tolerance test
- Diastolic blood pressure (mm Hg)
- Triceps skin fold thickness (mm)
- 2-Hour serum insulin (mu U/ml)
- Body mass index (weight in kg/(height in m)^2)
- Diabetes pedigree function
- Age (years)
- Class variable (0 or 1)
Missing Attribute Values: None
Class Distribution: (class value 1 is interpreted as “tested positive for diabetes”)
Class Value Number of instances 0 500 1 268
Brief statistical analysis:
Attribute number: Mean: Standard Deviation:
3.8 3.4120.9 32.069.1 19.420.5 16.079.8 115.232.0 7.90.5 0.333.2 11.8
Relabeled values in attribute ‘class’ From: 0 To: tested_negative
From: 1 To: tested_positive
Downloaded from openml.org.
Ensemble de données - return_X_y
fetch_openml renvoie un Bunch, un DataFrame ou X et y
Déséquilibre modéré (rapport inférieur à 3 ou 4)
Évaluation croisée
Ensemble d’entraînement et ensemble de test
Parfois appelée holdout method.
Recommandation: Allouer 80% de votre ensemble de données pour l’entraînement et réserver les 20% restants pour les tests.
Ensemble d’entraînement: Sous-ensemble de données utilisé pour entraîner votre modèle.
Ensemble de test: Sous-ensemble indépendant utilisé exclusivement lors de l’étape finale pour évaluer les performances du modèle.
Ensemble d’entraînement et ensemble de test
Erreur d’entraînement :
- En général, elle a tendance à être faible
- Obtenue en optimisant les algorithmes d’apprentissage pour minimiser l’erreur à travers l’ajustement des paramètres (par exemple, les poids)
Ensemble d’entraînement et ensemble de test
Erreur de généralisation : L’erreur observée lorsque le modèle est évalué sur des données nouvelles, non vues.
Ensemble d’entraînement et ensemble de test
Sous-apprentissage :
- Erreur d’entraînement élevée
- Le modèle est trop simple pour capturer les motifs sous-jacents
- Mauvaise performance à la fois sur les données d’entraînement et les nouvelles données
Sur-apprentissage :
- Faible erreur d’entraînement, mais erreur de généralisation élevée
- Le modèle capture le bruit ou des motifs non pertinents
- Mauvaise performance sur les nouvelles données non vues
Définition
La validation croisée (cross-validation) est une méthode utilisée pour évaluer et améliorer les performances des modèles d’apprentissage automatique.
Elle consiste à partitionner l’ensemble de données en plusieurs sous-ensembles, en entraînant le modèle sur certains sous-ensembles tout en le validant sur les autres.
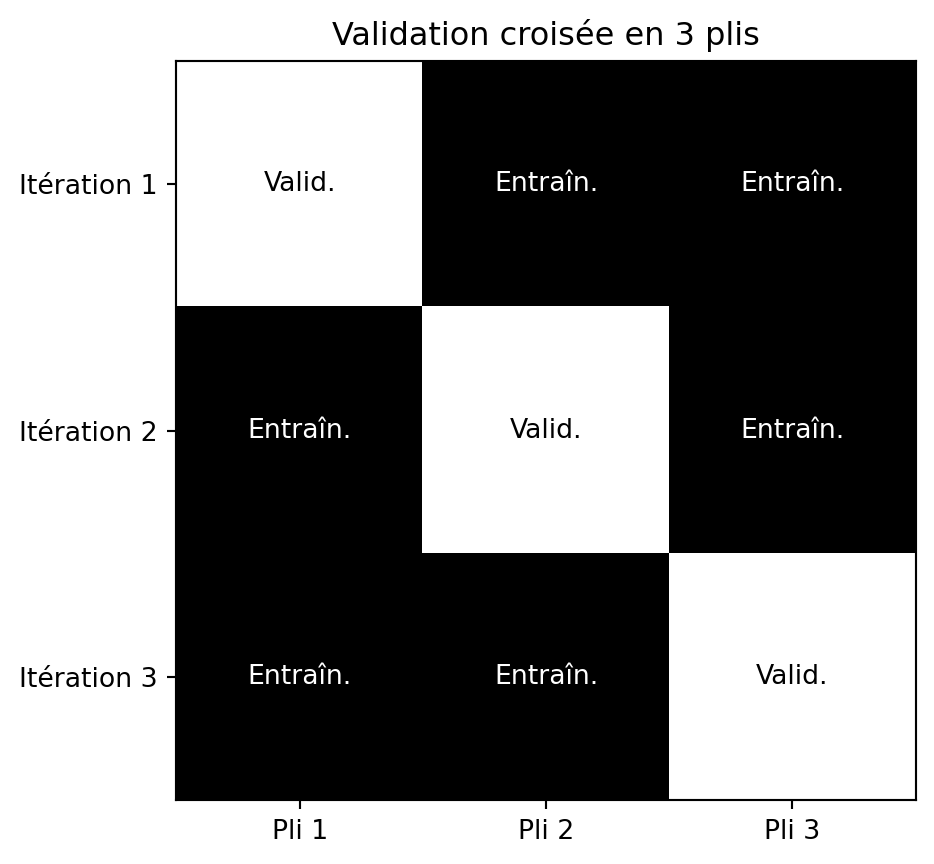
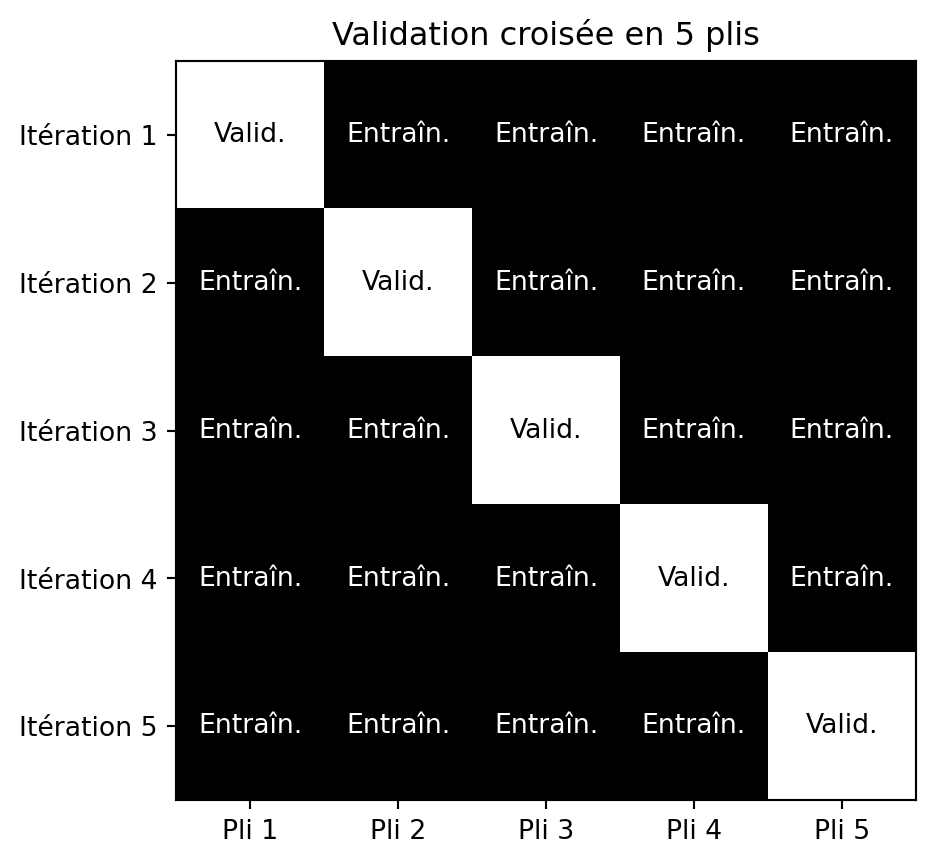
Validation croisée en \(k\) parties (k-fold)
- Diviser l’ensemble de données en \(k\) parties de taille égale (plis).
- Entraînement et validation :
- À chaque itération, un pli est utilisé comme ensemble de validation, les \(k\)-1 plis restants sont utilisés comme ensemble d’entraînement.
- Évaluation: Les performances du modèle sont évaluées à chaque itération, ce qui donne \(k\) mesures de performance.
- Agrégation: Les statistiques sont calculées à partir des \(k\) mesures de performance.
Validation croisée en 3 plis
Validation croisée en 5 plis
Évaluation de modèle plus fiable
- Estimation plus fiable des performances du modèle par rapport à une seule division entraînement-test.
- Réduit la variabilité associée à une seule division, conduisant à une évaluation plus stable et moins biaisée.
- Pour de grandes valeurs de \(k\), considérez la moyenne, variance, et intervalle de confiance.
Meilleure généralisation
- Aide à évaluer comment le modèle généralise à un ensemble de données indépendant.
- Garantit que les performances du modèle ne sont ni trop optimistes ni trop pessimistes en moyennant les résultats sur plusieurs plis.
Utilisation efficace des données
- Particulièrement bénéfique pour les petits ensembles de données, la validation croisée garantit que chaque point de données est utilisé à la fois pour l’entraînement et la validation.
- Cela maximise l’utilisation des données disponibles, conduisant à un entraînement de modèle plus précis et fiable.
Ajustement des hyperparamètres
- Couramment utilisé pendant l’ajustement des hyperparamètres, permettant de sélectionner les meilleurs paramètres du modèle en fonction de leurs performances sur plusieurs plis.
- Cela aide à identifier la configuration optimale qui équilibre biais et variance.
Défis
- Coût computationnel: Nécessite plusieurs entraînements de modèles.
- Leave-One-Out (LOO): Cas extrême où \(k = N\).
- Déséquilibre des classes: Les plis peuvent ne pas représenter correctement les classes minoritaires.
- Utilisez la validation croisée stratifiée pour maintenir les proportions de classes.
- Complexité: Implémentation sujette à des erreurs, en particulier pour la validation croisée imbriquée, les bootstrap ou l’intégration dans des pipelines plus complexes.
cross_val_score
from sklearn import tree
clf = tree.DecisionTreeClassifier()
from sklearn.model_selection import cross_val_score
clf_scores = cross_val_score(clf, X, y, cv=5)
print("\nScores:", clf_scores)
print(f"\nMoyenne: {clf_scores.mean():.2f}")
print(f"\nÉcart-type: {clf_scores.std():.2f}")
Scores: [0.71428571 0.66883117 0.71428571 0.79738562 0.73202614]
Moyenne: 0.73
Écart-type: 0.04Ajustement des hyperparamètres
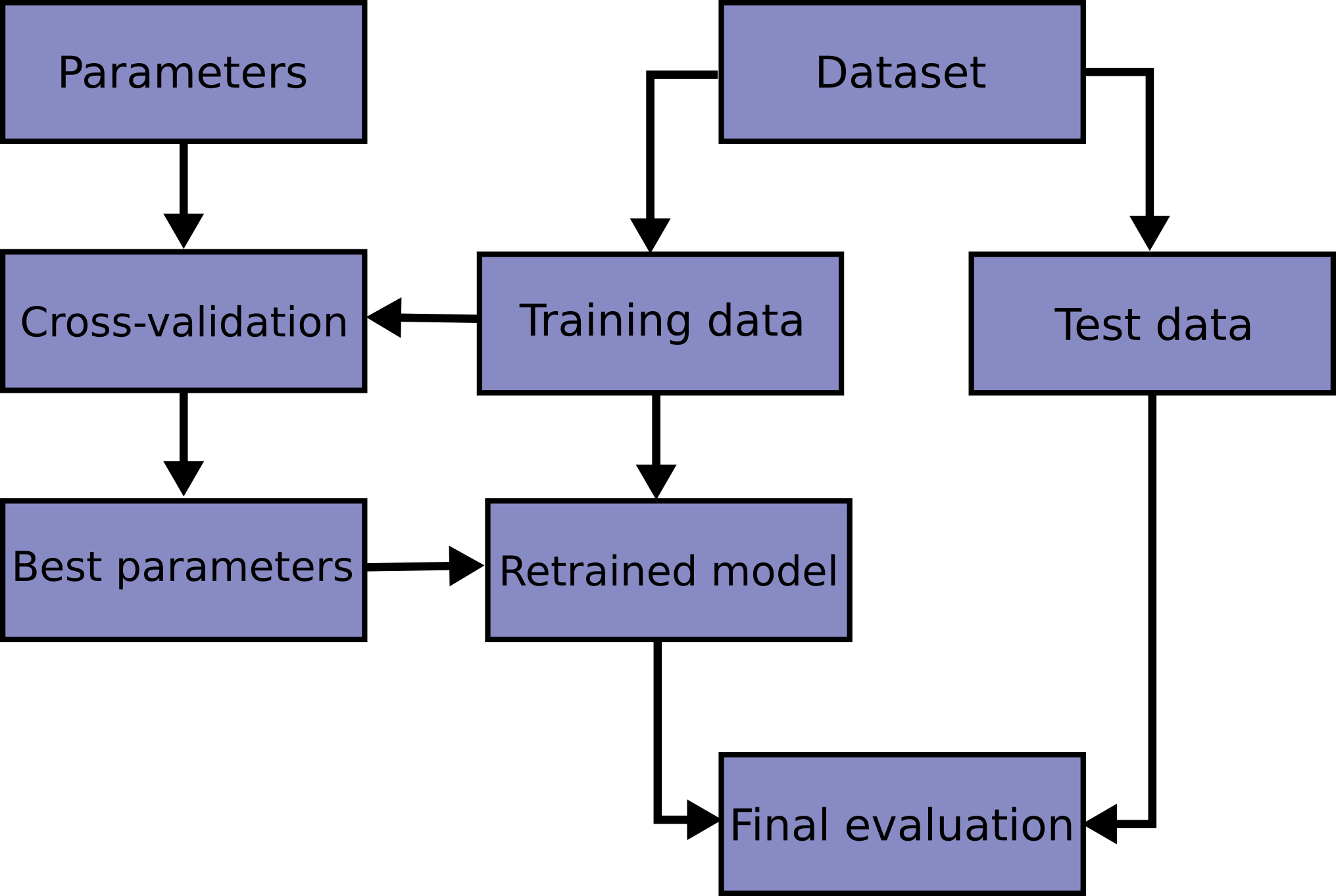
Flux de travail (Workflow)
Implémentation du flux de travail
Définition
Un hyperparamètre est une configuration externe au modèle qui est définie avant le processus d’apprentissage et qui régit le processus d’apprentissage, influençant les performances et la complexité du modèle.
Hyperparamètres - Arbre de décision
criterion:gini,entropy,log_loss, mesure la qualité d’une scission.max_depth: limite le nombre de niveaux dans l’arbre pour éviter le sur-apprentissage.
Hyperparamètres - Régression logistique
penalty:l1oul2, aide à éviter le sur-apprentissage.solver:liblinear,newton-cg,lbfgs,sag,saga.max_iter: nombre maximal d’itérations pour que les solveurs convergent.tol: critère d’arrêt, les petites valeurs signifient une plus grande précision.
Hyperparamètres - KNN
n_neighbors: nombre de voisins à utiliser pour les requêtes de \(k\)-voisins.weights:uniformoudistance, poids égal ou basé sur la distance.
Expérience : max_depth
for value in [3, 5, 7, None]:
clf = tree.DecisionTreeClassifier(max_depth=value)
clf_scores = cross_val_score(clf, X_train, y_train, cv=10)
print("\nmax_depth = ", value)
print(f"Moyenne: {clf_scores.mean():.2f}")
print(f"Écart-type: {clf_scores.std():.2f}")
max_depth = 3
Moyenne: 0.74
Écart-type: 0.04
max_depth = 5
Moyenne: 0.76
Écart-type: 0.04
max_depth = 7
Moyenne: 0.73
Écart-type: 0.04
max_depth = None
Moyenne: 0.71
Écart-type: 0.05Expérience : criterion
for value in ["gini", "entropy", "log_loss"]:
clf = tree.DecisionTreeClassifier(max_depth=5, criterion=value)
clf_scores = cross_val_score(clf, X_train, y_train, cv=10)
print("\ncriterion = ", value)
print(f"Moyenne: {clf_scores.mean():.2f}")
print(f"Écart-type: {clf_scores.std():.2f}")
criterion = gini
Moyenne: 0.76
Écart-type: 0.04
criterion = entropy
Moyenne: 0.75
Écart-type: 0.05
criterion = log_loss
Moyenne: 0.75
Écart-type: 0.05Expérience : n_neighbors
from sklearn.neighbors import KNeighborsClassifier
for value in range(1, 11):
clf = KNeighborsClassifier(n_neighbors=value)
clf_scores = cross_val_score(clf, X_train, y_train, cv=10)
print("\nn_neighbors = ", value)
print(f"Moyenne: {clf_scores.mean():.2f}")
print(f"Écart-type: {clf_scores.std():.2f}")Expérience : n_neighbors
n_neighbors = 1
Moyenne: 0.67
Écart-type: 0.05
n_neighbors = 2
Moyenne: 0.71
Écart-type: 0.03
n_neighbors = 3
Moyenne: 0.69
Écart-type: 0.05
n_neighbors = 4
Moyenne: 0.73
Écart-type: 0.03
n_neighbors = 5
Moyenne: 0.72
Écart-type: 0.03
n_neighbors = 6
Moyenne: 0.73
Écart-type: 0.05
n_neighbors = 7
Moyenne: 0.74
Écart-type: 0.04
n_neighbors = 8
Moyenne: 0.75
Écart-type: 0.04
n_neighbors = 9
Moyenne: 0.73
Écart-type: 0.05
n_neighbors = 10
Moyenne: 0.73
Écart-type: 0.04Expérience : weights
from sklearn.neighbors import KNeighborsClassifier
for value in ["uniform", "distance"]:
clf = KNeighborsClassifier(n_neighbors=5, weights=value)
clf_scores = cross_val_score(clf, X_train, y_train, cv=10)
print("\nweights = ", value)
print(f"Moyenne: {clf_scores.mean():.2f}")
print(f"Écart-type: {clf_scores.std():.2f}")
weights = uniform
Moyenne: 0.72
Écart-type: 0.03
weights = distance
Moyenne: 0.73
Écart-type: 0.04Recherche en grille (grid search)
De nombreux hyperparamètres nécessitent un ajustement
- Principal désavantage des algorithmes d’apprentissage automatique
L’exploration manuelle des combinaisons est fastidieuse
La recherche en grille est plus systématique
Énumérer toutes les combinaisons possibles d’hyperparamètres
Entraîner sur l’ensemble d’entraînement, évaluer sur l’ensemble de validation
GridSearchCV
from sklearn.model_selection import GridSearchCV
param_grid = [
{'max_depth': range(1, 10),
'criterion': ["gini", "entropy", "log_loss"]}
]
clf = tree.DecisionTreeClassifier()
grid_search = GridSearchCV(clf, param_grid, cv=5)
grid_search.fit(X_train, y_train)
(grid_search.best_params_, grid_search.best_score_)({'criterion': 'gini', 'max_depth': 5}, 0.7481910124074653)GridSearchCV
param_grid = [
{'n_neighbors': range(1, 16),
'weights': ["uniform", "distance"]}
]
clf = KNeighborsClassifier()
grid_search = GridSearchCV(clf, param_grid, cv=5)
grid_search.fit(X_train, y_train)
(grid_search.best_params_, grid_search.best_score_)({'n_neighbors': 14, 'weights': 'uniform'}, 0.7554165363361485)GridSearchCV
from sklearn.linear_model import LogisticRegression
# 2 * 5 * 5 * 3 = 150 tests!
param_grid = [
{'penalty': ["l1", "l2", None],
'solver' : ['liblinear', 'newton-cg', 'lbfgs', 'sag', 'saga'],
'max_iter' : [100, 200, 400, 800, 1600],
'tol' : [0.01, 0.001, 0.0001]}
]
clf = LogisticRegression()
grid_search = GridSearchCV(clf, param_grid, cv=5)
grid_search.fit(X_train, y_train)
(grid_search.best_params_, grid_search.best_score_)({'max_iter': 100, 'penalty': 'l2', 'solver': 'newton-cg', 'tol': 0.001},
0.7756646856427901)Recherche aléatoire
- Grand nombre de combinaisons (beaucoup d’hyperparamètres, beaucoup de valeurs)
- Utiliser RandomizedSearchCV :
- Fournir une liste de valeurs ou une distribution de probabilités pour les hyperparamètres
- Spécifier le nombre d’itérations (combinaisons à essayer)
- Temps d’exécution prévisible
Flux de travail
Enfin, nous procédons aux tests
clf = LogisticRegression(max_iter=100, penalty='l2', solver='newton-cg', tol=0.001)
clf.fit(X_train, y_train)
y_pred = clf.predict(X_test)
from sklearn.metrics import classification_report
print(classification_report(y_test, y_pred)) precision recall f1-score support
0 0.83 0.83 0.83 52
1 0.64 0.64 0.64 25
accuracy 0.77 77
macro avg 0.73 0.73 0.73 77
weighted avg 0.77 0.77 0.77 77
Prologue
Résumé
- Validation croisée et ajustement des hyperparamètres : Clé pour prévenir le surapprentissage et optimiser les performances.
Prochain cours
- Ingénierie de l’apprentissage automatique.
Références
Marcel Turcotte
École de science informatique et de génie électrique (SIGE)
Université d’Ottawa